Anleitung
Zur Anleitung gehts hier
Hintergrund Informationen - Background Information
Auf dieser Seite finden Sie jede Menge Informationen rund um das Thema Leuchtbakterien
Los gehts mit den Hintergrundinformationen zum Download als pdf:
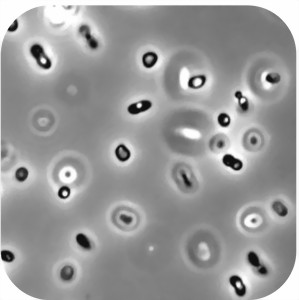
Abb. 1: Leuchtorganismen im Lichtmikroskop bei 400-facher Vergrößerung
Was sind Leuchtbakterien?
Oftmals sind es kommaförmige Mikroorganismen (lat.: Vibrionen) mit einer mittleren Größe von ca. 2 µm (0,002 mm). Diese haben die Fähigkeit ein blaugrünes Leuchten zu erzeugen. Dieses Licht ist teilweise so intensiv, dass die Umgebung erhellt werden kann. Dabei existieren sowohl solche Mikroorganismen, die einen Partner zum Leben brauchen (symbiontische Organismen), als auch solche, die allein leben können.
Leuchtbakterien sind weder Krankheitserreger noch verursachen sie Fäulnis.
Vorkommen:
Selbst in klarem Wasser ist es Sonnenlicht nicht möglich weiter als ca. 200 m tief vorzudringen. Die meisten Ozeane überschreiten diese Tiefe aber um ein Vielfaches und müssten daher stockdunkel sein. Aber eher das Gegenteil ist der Fall! Die Meere sind nämlich voll von Organismen die fähig sind Licht zu erzeugen. Dabei leben viele dieser Organismen mit Fischen (z.B. Merlan, Hering) zusammen. Bei dieser Art der Lebensform haben beide Partner Vorteile.
Die Leuchtorganismen bekommen ein Zuhause und eine geregelte Nahrungszufuhr, der Fisch eine Lampe. Mit der kann er dann im Dunkeln seine Nahrung finden und Räuber abschrecken.
Wie leuchten Bakterien?
Die Fähigkeit zu Leuchten ist abhängig vom Vorhandensein von Sauerstoff. Ein Enzym, die sogenannte Luciferase, wandelt den Sauerstoff in Wasser und Licht um. Dieses ist für uns als blaugrünes Leuchten (Biolumineszenz) erkennbar.
Ein weiterer Faktor, der nötig ist, damit die Organismen leuchten, ist ihre Anzahl. Nur wenn sich genügend Bakterien in der nächsten Umgebung befinden, können sie leuchten. Um festzustellen wie viele von ihnen da sind, benutzen sie ein komplexes System namens „Quorum Sensing“.
Nutzen der Leuchtbakterien:
Für den Symbiont (Fisch, Qualle, Krebs, Tintenfisch, …):
Zum einen dienen sie diesen als Lampe, damit sie in der Dunkelheit ihre Beute besser erkennen können. Der Anglerfisch zum Beispiel nutzt die Organismen als Köder, da seine leuchtende „Angel“ andere Beutetiere anlockt.
Zum anderen werden die Symbionten von den Organismen geschützt, da sie zur Abschreckung oder Tarnung eingesetzt werden.
Ein weiterer Nutzen ist die Erkennung des Geschlechts oder eines Paarungspartners. Dabei „kommunizieren“ die Fische, ähnlich einer Morsenachricht, über Lichtzeichen miteinander.
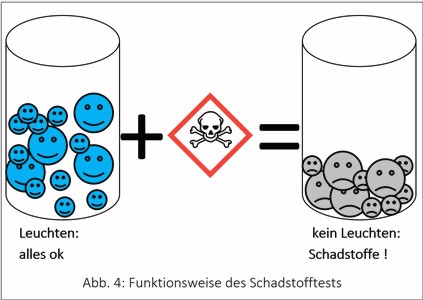
Abb. 4: Funktionsweise des Schadstofftests
Zur Qualitätssicherung von Wasser:
An der Leuchtintensität lässt sich die Güte des Wassers erkennen: Je besser die Wasserqualität, desto mehr Licht erzeugen die Organismen. Wird die Qualität schlechter, oder befinden sich Giftstoffe im Wasser, geht die Lichtausbeute zurück oder hört ganz auf.
Als Testverfahren für die Leistungsfähigkeit von Kläranlagen:
Über die Leuchtkraft der Bakterien kann die Reinigungs- und Entgiftungsleistung von Kläranlagen ermittelt werden.
Als Detektive für Luftschadstoffe:
Immer mehr finden biologische Tests mit Leuchtbakterien auch Anwendung bei der Detektion von Feinstäuben oder Emissionen aus Laserdruckern oder Kopierern.
Für weitere Informationen, Fragen, Bestellungen, Nachbestellungen oder individuelle Sets stehe ich Ihnen gerne zur Verfügung.
-------------------------------------------------------------------------------------------------
Ab hier ist es wohl eher etwas für die wissenschaftlich versierten Besucher der Seite
Der folgende Text enthält jede Menge Informationen rund um verschiedene Arten von Leuchtbakterien, deren Leuchtsysteme, deren Regulation auf genetischer Ebene und auch ein bisschen was zu symbiontischen Beziehungen zwischen Leuchtbakterien und anderen Organismen.
Viele (Bakterien) Zellen machen Licht - Biolumineszenz
Eine wissenschaftliche Betrachtung des Phänomens der leuchtenden Bakterien
Biologie der Leuchtbakterien
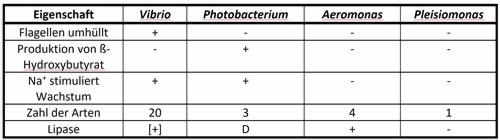
Früher wurden Leuchtbakterien in verschiedenen Gattungen zusammengefasst; heute sind folgende Gattungen/Arten anerkannt:
Familie Vibrionaceae: Gramnegative, gerade oder gekrümmte Stäbchen mit polaren Flagellen. Chemoorganotroph; alle können D-Glukose verwerten; fakultativ anaerob, fast alle Oxidase-positiv; Habitat: Wasser.
Alteromonas hanedai, ein strikt aerobes, freilebendes, marines Bakterium ist die einzige Art der Gattung Alteromonas, die Licht erzeugen kann (Luziferase basiert, ähnlich dem Vibrio-System)
Das Bakterium Xenorhabdus luminescens (Gattung der Familie Enterobacteriaceae), das fast ausschließlich innerhalb von Nematoden (hauptsächlich der Gattung Heterorhabditis) gefunden wird, zeigt ebenfalls Lumineszenz (die Zellen weisen eine Länge von 5 - 10 µm auf!).
Aus der Gattung Vibrio können Stämme der folgenden Arten blau-grünes Licht aussenden: Vibrio fischeri; Vibrio harveyi; Vibrio logei; und Vibrio splendidus.
Aus der Gattung Photobacterium können die Arten P. phosphoreum und P. leiognathi Licht aussenden, nicht jedoch P. angustum.
Die 4 Aeromonas-Arten (A. hydrophila, A. caviae, A. sobria, A. salmonicida), sowie Pleisiomonas shigelloides zeigen keine Biolumineszenz.
Alteromonas hanedai, ein strikt aerobes, freilebendes, marines Bakterium ist die einzige Art der Gattung Alteromonas, die Licht erzeugen kann (Luziferase ähnlich Vibrio-System)
Das Bakterium Xenorhabdus luminescens (Gattung der Familie Enterobacteriaceae), das fast ausschließlich innerhalb von Nematoden (der Gattung Heterorhabditis) gefunden wird zeigt ebenfalls Lumineszenz [Zellen 5 - 10 µm lang].
V. harveyi und V. fischeri können freilebend im Meerwasser vorkommen, mit einer Dichte von ca. 103 Zellen pro Liter. Bei der Symbiose kann V. fischeri bis zu 1010 Zellen pro ml erreichen (in den Leuchtorganen von Knochenfischen und Tintenfischen), während V. harveyi ein normaler Bewohner des Magen-Darm-Trakts von Fischen ist (bisher wurde keine Symbiose beobachtet). Biolumineszenz tritt nur auf, wenn hohe Zelldichten vorliegen, nicht z.B. bei freilebenden Bakterien da hierbei die notwendige Dichte nicht erreicht wird. Es sei angemerkt, dass der Unterschied im Leuchten vom reprimierten Zustand bis hin zum voll induzierten Zustand um den Faktor 104 gesteigert werden kann. Dies ist eine der höchsten bekannten Induktionsreaktionen.
Es ist zudem so, dass Bakterien, zumindest in den Leuchtorganen des Tintenfisches Euprymna scolopes nur langsam wachsen mit einer Verdopplungsrate von td = 4-5 Stunden; zum Vergleich erfolgt die Verdopplung der Zellen im Vollmedium ca. alle 30 Minuten. Im Vollmedium wenn die Zellen sehr gut wachsen, kann die Luziferase mehr als 5% des gesamten Proteins ausmachen. Unter solchen Bedingungen verbraucht das Leuchten dann allerdings auch mehr als 20% der Gesamtenergie der Bakterien. Das ist auch genau der Grund, warum das Leuchten auf den Nährmedienplatten nach Erreichen der vollen Leuchtkraft relativ schnell nachlässt, da extrem viel Energie verbraucht wird. Im Leuchtorgan, also in Symbiose, wird dagegen der Anteil am Gesamtenergieverbrauch auf ca. 0,001% geschätzt. Dies heißt natürlich auch, dass die Bakterien im Leuchtorgan wesentlich weniger intensiv leuchten als im Vollmedium. Allerdings kommen sie dort in extrem hoher Dichte vor. Beim Wachstum in Minimalmedium unter Laborbedingungen ergibt der Zusatz von Arginin drastisch verbessertes Leuchten, während höhere Salz Konzentrationen hemmend wirken. Daraus ist zu schließen, dass nicht nur die Zelldichte die Leuchtkraft reguliert sondern viele andere Faktoren ebenso einen Einfluss auf die Leuchtkraft haben. Glukose zum Beispiel reprimiert das Leuchten, was durch Zusatz von cAMP ins Medium wieder zumindest zeitweise aufgehoben werden kann. Für Vibrio harveyi wurde tatsächlich eine CRP-cAMP Bindestelle ca. 400 Nukleotide vor der Start-Stelle der mRNA gefunden. Allerdings regulieren hier auch noch andere Faktoren.
Leuchtbakterien können auch z.B. auf Fleisch wachsen, wie auf gepökeltem, da hoher Salzgehalt, Schinken. Nachdem diese Bakterien als harmlos gelten bzw. sogar zur Untersuchung der Wasserqualität verwendet werden, wurde das Leuchten von Fleisch als Indikator für Unverdorbenheit angesehen. In Berichten aus dem 19. Jahrhundert aus Militärhospitalen geht hervor, dass "leuchtende Wunden" ein Zeichen für einen guten Heilungsprozess waren. Interessanterweise wird meist Xenorhabdus luminescens in solchen Wunden gefunden, und dieses Bakterium ist ein Produzent von Antibiotika.
Vorteile der Symbiose:
Es wird spekuliert, dass die Bakterien den Vorteil einer zwar geringen, aber dafür kontinuierlichen Versorgung mit Nährstoffen in den Leuchtorganen haben. Gerade für marine Bakterien kann dies durchaus von Vorteil sein da vor allem offene Wasserflächen vom Nährstoffgehalt eher Wüsten ähneln. Es ist auch interessant zu wissen, dass der Inhalt der Leuchtorgane von den Fischen bewusst entleert werden kann, was als Hilfe zur Verbreitung der Bakterien gedeutet wird, in diesem Punkt ist sich die Wissenschaft aber nicht wirklich sicher. Für das mit Abstand am besten untersuchte Symbiosemodell Vibrio fischeri - Euprymna scolopes wurde nachgewiesen, dass der Tintenfisch tatsächlich das Bakterium mit zumindest 9 Aminosäuren versorgt, was für die Bakterien im Kontext der marinen Wüste natürlich einen enormen Wettbewerbsvorteil bietet.
Der Vorteil für die Fische wird im Leuchten gesehen; es sollen hier unterschiedliche Funktionen zugrunde liegen. Über Leuchtorgane sollen zum Teil:
- Feinde abgeschreckt werden
- Angehörige derselben Art erkannt werden
- Beleuchtung im Dunkeln der Tiefsee erzeugt werden
- potentielle Beute angelockt werden
- Tarnung, durch "Auflösung" von Körperschatten gegen den Himmel
Für den letzten Fall spricht z.B., dass bei einigen Fischen die Leuchtorgane so gestaltet sind, dass das Licht zunächst auf die Schwimmblase und den Darm fällt, die beide reflektierend sind, so dass auch ein kleines Leuchtorgan über diese Spiegel-Effekte schließlich den ganzen Fisch erstrahlen lassen kann. Hiervon ist sinnvollerweise nur die Unterseite betroffen, so dass Raubfische, welchen von unten nach oben blicken, den Umriss gegen den blauen Himmel nicht so leicht ausmachen können.
Die unterschiedlichen Organismen besitzen verschiedene, meist sehr spezifische Körperöffnungen, die den Bakterien den Zutritt zu den Leuchtorganen erlauben. Als weiterer interessanter Vertreter mit Leuchtorganen sei noch der japanische Tannenzapfenfisch genannt.
Gut leuchtende Bakterien emittieren ca. 104 Lichtquanten pro Zelle und Sekunde. Photorezeptoren reagieren auf minimal 10 Quanten. Unter Einbeziehung von "Verlusten" verschiedenster Art wurde kalkuliert, dass im Minimum 108 Zellen pro ml vorliegen sollten, um Photorezeptoren von z.B. Beuteorganismen der Fische anzusprechen. Mit der tatsächlichen Dichte von 1010 Zellen pro mL in den Leuchtorganen, ist dies sicher erreicht. Interessanterweise wird in Flüssigkultur im Labor das Leuchten ab ca. 107 Zellen pro ml induziert. Die Abhängigkeit des Leuchtens von der Zelldichte ist in mehrere Veröffentlichungen beschrieben. Bakterien der Gattungen Vibrio und Photobakterium können zusätzlich Proteine synthetisieren, die die Wellenlänge des emittierten Lichts von 490 nm durch Fluoreszenz modulieren. Dies kann sowohl in den niedrigeren als auch den länger welligen Spektralbereich führen.
Das Vibrio fischeri (inzwischen umbenannt in Alivibrio fischeri) System der Genregulation für die Biolumineszenz
Regulations-Region:
luxR und luxI werden divergent transkribiert, wobei eine intergene Region von ca. 200 bp existiert. Über entsprechende Deletions-Experimente und site-directed mutagenesis konnte festgestellt werden, dass ein 20 bp Palindrom, das kurz vor luxI liegt (ca. 40 bp), für die positive Autoregulation des "rechten" Operons von wesentlicher Bedeutung ist. Hier scheint also LuxR x Autoinduktor zu wirken und die Transkription zu erhöhen (bis um den Faktor 104). Vor diesem Palindrom liegt eine cis-aktive Sequenz, die das rechte Operon in Abwesenheit von LuxR abschaltet. Damit sind also zwei voneinander unabhängige Regulationen wirksam. Die Autoinduktion liegt darin begründet, dass LuxR x Autoinduktor das rechte Operon anschaltet, was gleichzeitig zur erhöhten Synthese des Autoinduktors führt.
Für Vibrio fischeri wurde inzwischen festgestellt, daß nicht nur ein, sondern zwei oder drei Autoinduktoren synthetisiert werden. AI1 ist OHHL, das vom luxI Genprodukt synthetisiert wird. Der Autoinduktor AI2, der als OHL identifiziert worden war, wird durch das Genprodukt ainS (Autoinduktor-Synthase) synthetisiert. Dabei ist ainS nicht homolog zu luxI. Durch die Erzeugung von luxI/ainS Doppelmutanten konnte gezeigt werden, daß von Vibrio fischeri kein weiterer Autoinduktor mehr gemacht wird. Dabei synthetisiert LuxI AI1 (OHHL) und auch - in wesentlich geringerer Menge - AI3 (HHL); AI2 (OHL) wird durch AinS erzeugt (Enzym-Mechanismus ähnlich LuxI). Regulation: AI2 hemmt kompetitiv die Assoziation von LuxR mit AI1. Also wirkt AI2 als Sicherheitsmechanismus, der eine Induktion des lux-Operons bei geringen bis intermediären Zelldichten verhindert wodurch das Leuchten erst bei Erreichen einer bestimmten Zelldichte zu beobachten ist. Ein ähnlicher Fall der Inhibierung eines Induktors liegt beim Sex Pheromon System von E. faecalis vor.
Symbiose zwischen Vibrio fischeri und Euprymna scolopes - Das mit Abstand am besten untersuchte Symbiose-Modell für Biolumineszenz.
Der ca. 5 cm lange Tintenfisch lebt im flachen warmen Meerwasser z.B. vor der Küste von Hawaii und versteckt sich tagsüber im Sand. In der Nacht geht er auf Futtersuche und beleuchtet seinen Körper über die Leuchtorgane zur „Schattenauflösung”. Die Tintenfische können leicht im Labor gezüchtet werden was deren Untersuchung enorm vereinfacht. Die Larven des Tintenfisches können dann gezielt mit Vibrio fischeri infiziert werden. Die Tintenfische stoßen jeden Tag ca. 90% der Bakterien aktiv während der Morgendämmerung aus dem Leuchtorgan aus. Während des Tages wachsen die Bakterien dann wieder zur „dichten Packung” in den Leuchtorganen hoch, ganz ähnlich eines kontinuierlichen Systems eines Chemostaten. Bakterien, die ausgestoßen wurden, können noch nicht infizierte Larven besiedeln und induzieren dort die Ausreifung der Leuchtorgane, wobei vielfältige Signale und sogar apoptotische Reaktionen eine Rolle spielen. Hier liegt eine Symbiose vor, der eine Koevolution beider Partner zugrunde liegt. Neuere Daten weisen darauf hin, dass die „erste Halbreaktion" der Luziferase, nämlich die Erzeugung von Enzym-Peroxid O2-, sowie weitere „Sauerstoff-Regulationen und Reaktionen" ganz wesentlich für die erfolgreiche Symbiose sind. In diesem sehr interessanten Spezialfall von Symbiose wurden anscheinend ursprüngliche Virulenzmechanismen eines Pathogens und Abwehrmechanismen des Wirts zu einer eindrucksvollen Symbiose kombiniert.
In den Tintenfischen Sepiola affinis und Sepiola robusta (beide leben im Mittelmeer) kommt ein Gemisch aus Vibrio fischeri und Vibrio logei vor. Damit wurde zum ersten Mal die Teilnahme der letzteren Bakterienart an einer Symbiose nachgewiesen und gleichzeitig gezeigt, dass die Leuchtorgane auch mehr als 1 Art enthalten können. Das heißt, in den Leuchtorganen befindet sich keine Reinkultur sondern vielmehr eine, biologischen Systemen ähnlichere, Mischkultur.
Das Vibrio harveyi System
Am Vibrio harveyi System ist nicht nur ein sondern zumindest zwei Autoinduktor(en) beteiligt. Zusätzlich reguliert durch eine phosphorylierungsabhängige Erkennung von „Umwelt-Signalen". Das System ist also wesentlich komplexer als das von Vibrio fischeri. Inzwischen ist auch klar, dass für Vibro harveyi kleine regulatorische RNA’s eine Rolle spielen.
Vergleiche der Systeme von Vibrio fischeri, Vibrio harveyi und gram positiven Bakterien
Beim Vergleich der verschiedenen Leuchtsysteme wird deutlich, dass das Vibrio fischeri-System das einfachste ist. Bei Vibrio harveyi wirken zwar auch Homoserin-Laktone, aber sie sind zunächst auf 2-Komponenten-Systeme verschaltet. Im Falle von Gram-pos. Bakterien wirken prozessierte Peptide, die wieder auf 2-Komponenten-Systeme verschaltet sind und dadurch wesentlich die Komplexität steigern.
Das Vibrio fischeri System
Hierbei ist es im Prinzip so, dass ein Signal-Molekül, der sog. Autoinduktor, das System anschaltet, wobei ein positiver Autoregulationskreis vorliegt. Mit anderen Worten, ein Signal wird von einem Rezeptor erkannt und führt u.a. zur erhöhten Synthese des Signals.
Zunächst aber kurz zum generellen, chemischen Mechanismus des Leuchtens. Im Prinzip wird dabei ein Aldehyd zur Säure oxidiert. Das entsprechende Enzym wird Luziferase genannt, die bei der Oxidation zur Erzeugung von Licht der Wellenlänge von 490 nm führt.
Die allgemeine Reaktion ist dabei:
FMNH2 + RCHO + O2 --> FMN + H2O + RCOOH + Licht
Das Enzym ist strikt FMNH2 abhängig. FMNH2 wird dabei selbst durch NADH2 reduziert, das natürliche Substrat ist allerdings Tetradekanal, ein C14-Aldehyd. Es handelt sich dabei um eine Zwei-Stufen-Reaktion, dazu aber etwas später mehr dazu. Es ist anzumerken, dass die entsprechenden Enzyme zur Substratsynthese natürlich für die Erzeugung dieses Substrats sorgen, wobei die Luziferase insbesondere bei niedrigen Substrat Konzentrationen praktisch nur dieses Substrat umsetzt. Andere Luziferasen, insbesondere die von Vibrio harveyi und X. luminescens setzen bei hohen Substrat Konzentrationen auch Nonal und Dekanal gut um. Bei hohen Substrat-Konzentrationen können die Luziferasen von Vibrio fischeri, P. phosphoreum und P. leiognathi sogar mit Dodekanal höhere Lichtausbeuten, als mit Tetradekanal ergeben.
Die Luziferase selbst ist ein Enzym mit einem Molekulargewicht von MW = 77 kDa, wobei die Untereinheiten ein MW von 40 und 37 kDa aufweisen. Die entsprechenden Gene luxA und luxB sind zueinander homolog und demzufolge in der Entwicklungsgeschichte wohl über Duplikation entstanden. Für alle bekannten Luziferasen liegt die Identität der Aminosäuren bei ca. 30%.
Neue Daten allerdings geben Anlass zu interessanten Spekulationen. Der Mechanismus der Luziferase wird mittlerweile so angenommen, dass in einem ersten O2-abhängigen Schritt ein unstabiles Flavin-Peroxid gebildet wird. Dieses kann in FMN + O2- zerfallen, wobei das entstehende H2O2 als Virulenzfaktor angenommen wird. Tatsächlich produziert zum Beispiel das Fisch-pathogene Vibrio salmonicida hohe Spiegel an Luziferase, allerdings findet Leuchten nur nach Zusatz eines Aldehyds statt. In der Natur leuchtet Vibrio salmonicida nicht, sondern greift Fischgewebe unter anderem durch H2O2 an. Eventuell wurde an die "Dunkelreaktion" der Luziferase eine Lichterzeugende Aldehyd-Oxidation angekoppelt. Zusätzlich kann noch argumentiert werden, dass ja im Leuchtorgan H2O2 als Abwehrmechanismus gegen bakterielle Besiedlung gebildet wird. Vibrio fischeri könnte nun durch die hohe Affinität der Luziferase zu O2 (im Bereich von nanomolar) diesen Abwehrmechanismus unterlaufen und sich so im Wirt etablieren.
Im Schatten dieser Überlegungen steht allerdings noch eine andere Überlegung: Kann die Fähigkeit zur Lichterzeugung eventuell mit dem Ausmaß der Photoreaktivierung des DNA-Repairs korreliert werden und somit dazu verwendet werden um DNA Schäden zu reparieren.
Im Folgenden wird die Organisation der lux-Gene und deren Funktion genauer betrachtet. Es muss allerdings im Hinterkopf behalten werden, dass die Analyse der Gene ursprünglich nicht die richtigen Ergebnisse lierferte, weil über die erfolgreiche Expression in E. coli zunächst einige Gene, die mit lux-Genen transkribiert werden, anfänglich übersehen wurden. Es ist demzufolge davon auszugehen, dass diese Gene in E. coli komplementiert werden können und somit trotzdem zur gewünschten Reaktion führten.
Die lux-Gene sind folgendermaßen organisiert: R stellt den Rezeptor (= Regulator) des Auto-Induktors dar, während I die entsprechende Synthase ist. Der Autoinduktor ist das N-(3-Oxohexanoyl)-L-Homoserin-Lakton. Die beiden Strukturgene für die Luziferase werden durch A und B kodiert, während C, D und E für Proteine kodieren, die das Substrat der Reaktion, nämlich den C14-Aldehyd synthetisieren. LuxD katalysiert eine Reaktion, in der aktivierte Fettsäuren von einem Carrier freigesetzt werden. Hierbei dient als Substrat insbesondere Tetradekanoyl-ACP (C14 – Acyl Carrier Protein), so dass also formuliert werden kann:
RCOX + HOH --> RCOOH + XH
mit X = ACP
LuxC + LuxE katalysieren die Erzeugung des Aldehyds nach:
RCOOH + ATP + NADPH --> NADP + AMP + PPi + RCHO
Für P. phosphoreum wurde nachgewiesen, dass alle drei Enzyme miteinander komplexiert sind, wobei ein zentrales Tetramer der Reduktase (4 x 54 kDa LuxC) umgeben wird von 4 Synthetase-Einheiten (4 x 42 kDa LuxE) an die wieder je eine Transferase gekoppelt ist (4 x 33 kDa LuxD). Damit hat der Gesamtprotein-Komplex eine Größe von ca. 5 x 102 kD.
Für LuxG ist eine Funktion als FMN/NADH2-Reduktase angenommen. Es ist in X. luminescens nicht vorhanden und wird auch in E. coli nicht für funktionierende Lumineszenz gebraucht, da dort eine andere Reduktase vorhanden ist. Deshalb wurde früher spekuliert, dass LuxG eventuell für die Adaptation an "Salzreiche-Habitate" nötig ist. Zumindest ein weiteres Gen, luxH, wurde inzwischen in V. harveyi als Teil des Operons gefunden, allerdings nicht in Vibrio fischeri.
Seit 2007 ist auch bekannt, dass der Sauerstoff-Sensor ArcA das Leuchtsystem reguliert. Dies macht auch Sinn, vor allem wenn man bedenkt, dass bis zu 10-20% der Gesamtenergie der Zelle von gut leuchtenden Bakterien für die „Lichtproduktion“ verbraucht wird. Wie wichtig das Ganze ist, kann daraus ermessen werden, dass die Bakterien Energie über aerobe Atmung gewinnen, aber bei Leuchten verbrauchen sie Sauerstoff, ATP und Reduktionsäquivalente, was einen enormen Energieverbrauch darstellt.
Verschiedene Daten haben gezeigt, dass LuxI eine Acyl-Homoserinlakton-Synthase ist. Demzufolge wird über ein Hexanoyl-AcylCarrierProtein das Hexanoyl auf eine (Cystein-) SH-Gruppe von LuxI übertragen. Im Anschluss wird S-Adenosyl-Methionin gebunden. Dann wird Hexanoyl auf SAM gebunden, und anschließend Methyl-S-Adenosin abgespalten. In einer Laktonisierungs-Reaktion wird schließlich das Hexanoyl-Homoserinlakton freigesetzt. Die regulation erfolgt über eine Regulations-Region:
luxR und luxI werden divergent transkribiert, wobei eine intergene Region von ca. 200 bp existiert. Über entsprechende Deletions-Experimente und site-directed mutagenesis wurde festgestellt, dass ein 20 bp Palindrom, das kurz vor luxI liegt (ca. 40 bp), für die positive Autoregulation des "rechten" Operons von wesentlicher Bedeutung ist. Hier bindet also LUX-R x Autoinduktor als Dimer und erhöht so die Transkription um den Faktor 104. LuxR x Autoinduktor bindet zum einen an die RNAP im Bereich von ßß’ und zum anderen an die C-terminale Domäne. Vor dem als „lux-box” bezeichneten Palindrom scheint eine cis-aktive Sequenz zu liegen, die das rechte Operon in Abwesenheit von LuxR abschaltet. Damit sind dann also zwei voneinander unabhängige Regulationen wirksam. Die Autoinduktion liegt also darin begründet, dass LuxR x Autoinduktor das rechte Operon anschaltet, was gleichzeitig zur erhöhten Synthese des Autoinduktors führt.
Ein solches Modell der positiven Rückkopplung kann erklären, weshalb bereits ein Molekül des Autoinduktors pro Zelle das gesamte System induzieren kann - es findet ja laufend eine Induktion der Induktor-Synthese statt. Andrerseits liegt in einem solchen positiven autoregulatorischen System auch eine Gefahr: es muss gut abschaltbar sein, sonst würde es - über seine eigene, permanente Induktion - die Zelle anderer wichtiger Synthesekapazität berauben. Insofern ist also eine negative Regulation von LuxR auf seine Synthese durchaus sinnvoll. Es könnte gut sein, dass das V. fischeri System eher eine Ausnahme für die Regulation darstellt, und in den anderen Systemen noch weitere Regulatoren wirken. Insbesondere in einem solchen Fall macht natürlich der positive Autoregulationskreis Sinn.
Es wurde gezeigt, dass LuxR fast absolut spezifisch für seinen passenden Autoinduktor ist. Dies erklärt, weshalb Kulturüberstand von Vibrio fischeri nicht das Vibrio harveyi System induzieren kann und umgekehrt. Diese Spezifität ist allerdings nicht 100%-ig. Das war aber schon seit langem bekannt da schon vor 1950 festgestellt wurde, dass Kulturüberstand bestimmter Bakterien durchaus das V. fischeri System anschalten kann. Es muss auch noch erwähnt werden, dass der Autoinduktor frei membrangängig ist und sich deshalb keine großen Unterschiede in der Konzentration innerhalb der Zellen zum Medium ergeben.
LuxR von V. fischeri ist allerdings ein Protein, das an der Cytoplasma-Membran gebunden ist, obwohl aus der Sequenz keine Membran durchspannende Helix vorhergesagt wird. Es muss betont werden, dass der negative auto-regulatorische Effekt von LuxR auf luxR in Domäne 1 liegt. Domäne 2 bewirkt die Bindung des Autoinduktors, während Domäne 3 für eine Multimerisierung des Proteins verantwortlich ist. LuxR besitzt ein sog. Aktivator-Modul, das für die Interaktion mit der zu regulierenden DNA wichtig ist. Domäne 4, die in diesem Aktivator-Modul liegt, stellt dabei ein Helix-Turn-Helix Motiv dar, das für DNA-Bindung erforderlich ist. Die Domäne 5 schließlich ist für die eigentliche Aktivierung der Transkription verantwortlich. Damit ist LuxR ein Mitglied einer Familie an Regulatoren, die z.B. LAS-R (Pseudomonas aeruginosa) und TraR (Agrobacterium tumefaciens) enthalten. Ebenso zeigen RhiR aus Rhizobium leguminosarum und SdiA aus E. coli Ähnlichkeiten mit LuxR. LasR, TraT, RhiR und SdiA zeigen jeweils über die gesamte Länge des Proteins Ähnlichkeiten zu LuxR. Zusätzlich gehören dieser Familie noch FixJ (Rhizobium meliloti) und NarL (E. coli) an, die selbst wieder Angehörige von Zwei-Komponenten-Regulations Systemen sind.
Autoinduktor-Analoge, die im HSL-Ring verändert sind, binden +/- nicht an LuxR, während Änderungen in der Seitenkette z.T. noch Bindung erlauben, und so zum An- oder auch Abschalten des Systems führen. Allerdings ist die beste Bindung für das homologe Paar LuxR x Hexanoyl-HSL nachgewiesen worden.
Nachdem auch in den später gezeigten Daten nicht darauf eingegangen wird, sei hier nur ganz kurz erwähnt, dass das Fisch-pathogene Bakterium Vibrio anguillarum ebenfalls einen Autoinduktor bildet, nämlich 3-Oxodecanoyl-HSL. Die vanI/R Gene zeigen Ähnlichkeiten zu denen von V. fischeri.
Das Vibrio harveyi System
Es wurde ja schon gesagt, dass das V. harveyi System wesentlich komplexer ist als das V. fischeri System. Insbesondere liegen hier mehr lux-Gene vor und es sind zusätzliche Regulations-Mechanismen eingeschaltet. Es sei auch darauf hingewiesen, dass die Lumineszenzfarbe durchaus nicht immer „gelbgrün” sein muss.
Bei V. harveyi ist die Organisation der „Leuchtgene" anders als bei V. fischeri: es existiert zwar ein luxCDABEGH-Operon, aber in der Nähe liegen keine Gene, die zu luxR und luxI (von V. fischeri) homolog sind. Es wurden auch im gesamten Chromosom keine homologen Gene gefunden. Andrerseits gibt es tatsächlich ein LuxR Protein, das für die Aktivierung des lux-Operons wichtig ist, aber eben keine Ähnlichkeiten zum LuxR von V. fischeri zeigt. Es ist außerdem klar, dass das Gen für dieses V. harveyi LuxR Protein nicht benachbart zum lux-Operon liegt. Die Funktion der zusätzlichen lux-Gene luxGH ist relativ unklar. Vermutet wird, dass diese evtl. für die Synthese des reduzierten Flavinsubstrats gebraucht werden.
In Bezug auf die Regulation ist wesentlich mehr bekannt. Im V. harveyi System wirkt nicht nur ein, sondern zwei Autoregulatoren; hier ist zusätzlich noch ein Phosphorylierungs-System beteiligt. Es existiert nämlich ein übergeordneter Repressor für das lux-Operon, der von luxO kodiert wird. LuxO reprimiert das System, wobei seine Phosphorylierung die Repressorfunktion ermöglicht. Nicht phosporyliertes LuxO wirkt nicht als Repressor. LuxO selbst wird von einer Kinase phosphoryliert, die von luxU kodiert wird. Die Membran-gebundenen Proteine LuxN und LuxQ wirken dabei als Sensoren, die die Konzentration der Autoinduktoren AI-1 und AI-2 feststellen. LuxN (und O) selbst hat Sequenz-Ähnlichkeiten mit Histidin-Kinasen und den Sensor-Proteinen von Zwei-Komponenten Systemen. D.h., dass hier beide Proteine der Zwei-Komponenten-Systeme in LuxN (und O) funktionell zusammengefasst sind (dies ist z.B. auch im Falle von FrzE aus Myxococcus xanthus). Bei niedrigen Zelldichten (= ohne AI-1) wirkt LuxN als Kinase, die im Endeffekt das Phosphat über LuxU auf LuxO überträgt; phosphoryliertes LuxO bewirkt aber zusammen mit dem alternativen Sigma-Faktor die Expression eines unbekannten Repressors X. Bei hohen Zelldichten wird ja AI-1 gemacht und die Kinase-Aktivität von LuxN fällt aus. Die Phosphatase-Aktivität dagegen bleibt erhalten, so dass nun LuxO dephosphoryliert wird und so die lux-Gene über LuxR angeschaltet werden.
Die Bildung des Autoinduktors AI-1 (= 3-Hydroxybutanoyl-HSL) wird durch die Proteine LuxL und LuxM bewirkt, die allerdings keinerlei Ähnlichkeiten zu LuxI von V. fischeri zeigen. luxLMN liegen bei V. harveyi in einem Operon. Die Struktur des Autoinduktors AI-2 ist seit kurzem bekannt und zeigt, dass nicht nur E. coli und S. typhimurium eine AI-2 ähnliche Substanz produzieren, sondern auch Gram-positive Bakterien.
Im V. harveyi System sind also zwei Autoinduktoren wirksam; zusätzlich ist eine Phosphorylierungs-abhängige Repressor-Ebene und ein neuartiger Aktivator eingebaut. Das System ist also wesentlich komplexer als das von V. fischeri. CRP dagegen braucht cAMP um dann als Aktivator zu fungieren (Ausschalten des Gens gibt 1.000-fach weniger Licht). Beide Proteine binden in der Promotor-Region des lux-Operons, sodass also nicht nur die Zelldichte (AI-1 und AI-2) reguliert, sondern auch physiologische Zustände.
Ende 2004 nun wurde sogar noch ein drittes QS-System von V. harveyi identifiziert. Dieses wirkt parallel zu den anderen beiden Systemen LuxM-N und LuxS-PQ; es wurde als Cqs-System bezeichnet. Dies kommt daher, dass die Produktion mit einem Vibrio cholerae Stamm identifiziert wurde, der auf seinen Autoinduktor CAI-1 reagiert und dessen Sensor mit CqsS bezeichnet wurde. Tatsächlich fand man auch im Genom von V. harveyi die entsprechenden cqsA und cqsS Gene. Dabei wirkt das Cqs-System bei wesentlich geringeren Zelldichten, als die anderen beiden Systeme.
Es sollte auch erwähnt werden, dass V. harveyi ein Pathogen für Garnelen sein kann und in kommerziellen Zuchtbetrieben sehr große Probleme bereiten kann, bis hin zum Totalausfall der Larven. Hier ist momentan nur klar, dass das Leuchten und die Produktion eines extrazellulären Toxins über die lux-Systeme reguliert werden.
Neuere Daten zu V. fischeri
Für V. fischeri wurde gefunden, dass nicht nur ein, sondern zwei bzw. drei Autoinduktoren existieren. AI1 ist OHHL, das über das luxI Genprodukt synthetisiert wird. Bei der Sequenzierung stellte es sich nun heraus, daß zwei Gene ainS und ainR direkt hintereinander in der gleichen Transkriptionsrichtung im V. fischeri Genom vorkommen. In E. coli ist ainS ausreichend für die Synthese von AI-2. Der hierbei wesentliche Befund ist, daß ainS nicht homolog zu luxI ist. Die C-terminale Hälfte des Proteins AinS zeigt vielmehr Homologien zu LuxM aus V. harveyi. Das nachgeschaltete Protein AinR zeigt Homologien zu LuxN aus V. harveyi. Damit sind nun also doch Homologien für diese beiden Arten festellbar, wobei sich diese ergeben für das AI2 - System von V. fischeri mit dem luxMN-System von V. harveyi. Letzteres wird ja benötigt, um den HAI1/Sensor-Komplex zu bilden, der dann wieder den Repressor LuxO inaktiviert. Durch die Erzeugung von luxI/ainS Doppelmutanten konnte gezeigt werden, dass für P. fischeri kein weiterer Autoinduktor mehr gemacht wird. Damit synthetisiert also LuxI AI1(OHHL) und auch - in wesentlich geringerer Menge - AI3 (HHL); AI2 (OHL) wird durch AinS erzeugt. Der enzymatische Mechanismus ist für LuxI und AinS sehr ähnlich. Die Regulation durch AI2 sieht nun so aus, dass AI2 kompetitiv die Assoziation von LuxR mit AI1 hemmt. Dies geschieht so, dass AI2 x LuxR eine wesentlich geringere Affinität zum luxICDABEG-Operon hat als AI1 x LuxR. Demzufolge wirkt dann AI2 als Sicherheitsmechanismus, der eine Induktion des lux-Operons bei geringen bis intermediären Zelldichten verhindert.
Inzwischen wurde auch ein litR-Gen in V. fischeri gefunden, dessen Genprodukt zu ca. 60% mit LuxR aus V. harveyi identisch ist. Tatsächlich handelt es sich dabei auch um einen Regulator, der die Transkription des V. fischeri luxR-Gens reguliert – wird litR inaktiviert, so findet man nur noch ca. 20% Leuchtkraft in den entsprechenden Stämmen.
Symbiose zwischen V. fischeri und Euprymna scolopes
Dies ist das mit Abstand am besten untersuchte Symbiose-Modell für Biolumineszenz. Es gibt zwar wesentlich spektakulärer leuchtende Tintenfische als E. scolopes, für diese ist aber kaum etwas bekannt. Der ca. 5 cm lange Tintenfisch E. scolopes lebt im flachen warmen Meerwasser z.B. vor der Küste von Hawaii und versteckt sich tagsüber im Sand. In der Nacht geht er auf Futtersuche und beleuchtet seinen Körper über die Leuchtorgane zur „Schattenauflösung”, wobei der Tintenfisch die Leuchtstärke je nach der Helligkeit der Mondnacht regeln kann.
Die Tintenfische können leicht im Labor gezogen werden. Nach dem Schlüpfen sind die Larven noch unbesiedelt; sie können leicht gezielt mit V. fischeri infiziert werden. Ein Vorteil dieses Systems ist, dass die Tintenfische jeden Tag ca. 90% der Bakterien aktiv während der Morgendämmerung aus dem Leuchtorgan ausstoßen. Während des Tages wachsen die Bakterien dann wieder zur „dichten Packung” in den Leuchtorganen hoch. Bakterien, die ausgestoßen wurden, können noch nicht infizierte Larven besiedeln. Tatsächlich ist auch Wasser, das aus Regionen stammt, in denen viele Tintenfische leben, besonders infektiös. Die Besiedlung durch Bakterien induziert die Ausreifung der Leuchtorgane, wobei sogar apoptotische Reaktionen eine Rolle spielen. Zwar können alle symbiontischen Leuchtbakterien-Arten den Tintenfisch infizieren, allerdings zeigen sich in Mischexperimenten immer die Stämme besonders gute Besiedlung, die aus Symbiosen mit dem Tintenfisch stammen. Insgesamt gesehen ist klar, dass hier eine Symbiose vorliegt, der eine Koevolution beider Partner zugrunde liegt.
Gerade in den letzten Jahren wurde dieses System sehr intensiv untersucht, wobei die Vielfalt der wechselseitigen Interaktion klar wurde, d.h. es findet eine Erkennung beider Partner statt. Einige Zahlenwerte:
Im Meerwasser ca. 106 Bakterien/mL; ca. 0,02 V. fischeri Zellen/mL im freien Meerwasser, aber ca. 2/mL in den flachen Buchten in denen Euprymna scolopes vorkommt. Nach einigen Untersuchungen sogar 600 V. fischeri Zellen/mL im Symbiose-Biotop, aber selbst dann bestenfalls 0,1% aller vorkommenden Bakterien. Frisch geschlüpfte Larven von Euprymna tragen erstmal keine Symbionten, haben noch kein voll differenziertes Leuchtorgan und sind recht klein. Sie ventilieren ca. 3 – 4-mal pro Sekunde und bewegen dabei nur ca. 1,5 µl an Meerwasser. Wenn dieses ca. 500 V. fischeri Zellen pro mL enthält dann wird ca. 1 Zelle alle 0,3 Sekunden am Leuchtorgan vorbeigespült. Die Eintrittspforte ins Leuchtorgan besteht aus 6 Poren mit je ca. 15 µm2 Öffnungsfläche, d.h. ein zufälliger Kontakt könnte nie funktionieren. Bei gezielten Infektionsversuchen zeigte sich, dass insgesamt ca. 250 Bakterien pro Tintenfisch genügen, um eine effektive Besiedlung zu erreichen.
Wie wird aber die hohe Spezifität erreicht? Dazu ist es auch wichtig, die Morphologie des Leuchtorgans zu verstehen. Meist ist es als schwarzer Fleck durch die Larve hindurch zu erkennen. Tatsächlich liegen hier übereinander / nebeneinander der Tintensack, Reflektoren und 2 x 3 Divertikel, sowie die eigentlichen 6 internen Crypten, die als Einstülpungen im Mantelbereich zu bezeichnen sind. Beim Ventilieren wird nun das Wasser über den Trichter wieder ausgestoßen und fließt dabei am Cilienfeld vorbei. An diesem befinden sich links und rechts je 2 bewegliche „Lappen" an deren Grund die jeweils 3 (also insg. 6) Eintrittsporen angelegt sind. Von den Poren aus geht ein Gang (= Ductus) zu den jeweiligen Crypten, von der größten der jeweils 3 Crypten ziehen sich dann 3 Divertikel als "Blindsäcke" weiter. Während V. fischeri in den Crypten noch relativ normal aussieht, d.h. begeißelt und bis 2 µm lang, sind die Bakterien in den Divertikeln zu unbeweglichen, nur noch 0,5 µm langen Zellen umgebildet.
Nach der Besiedlung ergeben sich dramatische morphologische Veränderungen der Crypten: Die Cryptenzellen schwellen an und bilden einen sehr dichten Saum an Mikrovilli aus, in dem sich die Bakterien ansiedeln.
Allerdings befinden sich in der Crypte auch Makrophagen, die tatsächlich auch Bakterien in der Crypte verdauen können. Es wurde ja schon angesprochen, dass die Bakterien täglich ausgestoßen werden. Aus einem der Ducti wird auf der rechten Seite das hochgewachsene Gemisch aus Bakterien, Wirtszellen und Membranvesikeln ausgestoßen. Eine geringe Zahl an Bakterien bleibt immer in der Crypte/Divertikel zurück, die dann tagsüber wieder zu hoher Zelldichte heranwachsen – stationäre Zellen hören ja mit der Zeit auf zu leuchten, so dass auf diese Weise sichergestellt wird, dass immer Bakterien in der richtigen Wachstumsphase vorhanden sind.
Es wird also klar, dass hier tiefgreifende morphologische Änderungen im Tintenfisch induziert werden, dafür sind z.T. auch apoptotische Effekte wichtig. Ende 2004 wurde nun ein hierfür wichtiger Faktor identifiziert. Das Bakterium scheidet aktiv einen Baustein des Mureins aus, der als TCT bezeichnet wird. TCT hat seinen Namen erhalten als Tracheal Cytotoxin, also einem Faktor der verantwortlich ist für massive Gewebsschädigungen, wie sie bei Keuchhusten und Gonorrhoe gefunden werden – tatsächlich ist bisher auch nur für Bordetella pertussis und für Neisseria gonorrhoeae die Produktion von TCT berichtet worden. Zusatz des Murein-Bestandteils (= NAz-Glu – NAz-Mur – Ala-Glu-DAP-Ala) in Konzentrationen von < 1 µM erzeugt die für V. fischeri typischen morphologischen Veränderungen. TCT erzeugt bei Keuchhusten und Gonorrhoe den Verlust von Cilien-Zellen im Atemtrakt bzw. den Eileitern, und verstärkt dramatisch den Effekt von LPS auf die Produktion von imflammatorischen Cytokinen.
V. fischeri erkennt bei der Besiedlung also den Schleim, der von der Pore ausfließt, und wächst zu Minikolonien heran, die in Ductus und Crypte einwandern. Dabei müssen die Bakterien im Ductus der Bewegung von Cilien entgegenkriechen, die eigentlich der Ausschleusung von Bakterien dienen. In der Crypte müssen die Bakterien den Makrophagen „entkommen" – nachgewiesen ist, dass andere Bakterien über die Makrophagen getötet werden. Die Bakterien wachsen mit td von 20 min auf ca. 105 Zellen hoch (10 – 12 Stunden). Das Volumen einer Crypte ist ca. 750 pL, d.h. die Konzentration im Leuchtorgan liegt bei ca. 1011 Zellen/mL. Die Bakterien wachsen danach nur noch mit td = 5,5 h; sie werden aber auch bald wieder ausgeschieden. Wenn die Crypte "voll ist", beginnt das Leuchten – lux-Mutanten können nicht richtig infizieren (über lux-System wohl nicht nur das Leuchten reguliert, das bis zu 20% der Zell-Energie verbrauchen kann). Klar ist, dass im Leuchtorgan hohe Konzentrationen an Aminosäuren vorkommen. Evtl. dient auch noch eine proteolytische Mucinase für die weitere Ernährung. Innerhalb 2 – 4 Tagen schwellen die Epithelzellen der Crypten an, die starke Vermehrung der Mikrovilli wird induziert, so daß zum Schluss alle Bakterien mit Wirtszellen in engem Kontakt sind.
Zum „Schleim" den Euprymna bildet ist noch nicht zu viel bekannt. Was allerdings inzwischen bekannt ist:
- Er wird erst ausgebildet, wenn im Umgebungswasser Gram-neg. Zellen vorhanden sind
- 2 Komponenten des Schleims sind N-Acetyl-Neuraminsäure und N-Acety-Galaktosamin; als chemoattraktive Komponenten wurden inzwischen Serin, Nucleoside und N-Azetyl-Neuraminsäure identifiziert
- Bei Misch-Infektions-Experimenten wurde klar, dass V. fischeri andere (ähnliche, z.T. leuchtende) Bakterien wie Vibrio parahaemolyticus oder Photobacterium leiognathi auskompetitiert. Dabei scheint wohl der Effekt darauf zu beruhen, dass diese potentiellen „Konkurrenten“ nicht etwa bekämpft werden, sondern einfach den Schleim nicht als chemoattraktiv empfinden
- V. fischeri muss aktiv durch den Schleim zur Pore wandern. Die Flagellen des Bakteriums sind aus mehreren Flagellinen aufgebaut (insg. 6 Gene bekannt), wobei FlgA essentiell für die Besiedlung ist
- Auffallend ist, daß ca. 2 – 4 Stunden vergehen, bis die Bakterien nach dem ersten Kontakt mit dem Schleim zur Pore hinwandern. Es kann sein, dass hier noch ein Signalaustausch statt findet
Man weiß über Proteom-Untersuchungen inzwischen auch, dass sich für das Leuchtorgan in den ersten 24 Stunden nach Erstinfektion relativ wenig tut. Nach 2 Tagen dagegen ergeben sich deutlich Unterschiede; weitere sind bis zu 4 Tagen nach Erstbesiedlung sichtbar. Es dauert auch ungefähr 4 Tage, bis schließlich ein "fertiges" Leuchtorgan im jungen Tintenfisch vorliegt. Im Bakterienbereich ist z.B. klar, dass zumindest ein 2-Komponentensystem existiert, das für die Besiedlung wichtig ist – welches Signal erkannt wird, ist allerdings unklar. Man ist hier momentan sehr aktiv auf der Suche nach entsprechenden Faktoren z.B. auf der Oberfläche von V. fischeri welche die spezifische Besiedlung ermöglichen.
Wie spezifisch diese Interaktionen sind, kann daran erkannt werden, dass:
- Endogene Symbionten immer von außen zugesetzte V. fischeri Stämme auskompetitieren
- Besiedlung des Leuchtorgans nur mit V. fischeri möglich ist – andere Bakterien können zwar reingelangen, aber sie werden eliminiert
Luziferase-Reaktion und "Sauerstoff-Regulationen"
Neue Daten lassen interessante Spekulationen zu: der Mechanismus der Luziferase ist nämlich so, dass in einem ersten O2-abhängigen Schritt ein unstabiles Flavin-Peroxid gebildet wird. Dieses kann in FMN + O2- zerfallen, wobei das entstehende H2O2 als Virulenzfaktor angesehen wird. Die Bindung von O2 ist dabei außerordentlich gut (KM im nMolaren Bereich!), so dass das fakultativ anaerobe Bakterium seine Umgebung +/- Sauerstofffrei halten kann. Wenn nun kein Aldehyd zur weiteren Reaktion zur Verfügung steht, dann entsteht tatsächlich O2-; das Superoxidanion könnte theoretisch als Angriffsmechanismus gegen die Eukaryonten gebraucht werden. V. cholerae produziert z.B. tatsächlich einen Autoinduktor.
Zusätzlich ist aber auch zu sehen, dass ja bei der Symbiose noch weitere, "Sauerstoff-Regulationen" greifen:
- Der Tintenfisch Euprymna produziert ja eine sog. Haloperoxidase, die sowohl auf seinen Kiemen, wie auch im Leuchtorgan gefunden wird. Die Haloperoxidase erzeugt hypochlorige Säure (HOCl) als Schutz gegen pathogene Bakterien. Kurz nach der Besiedlung des Leuchtorgans wird der Spiegel an mRNA für Haloperoxidase drastisch herabgesetzt
- V. fischeri eine sehr potente Katalase besitzt, die im Periplasma lokalisiert ist. Damit könnte evtl. entstehendes H2O2 weggefangen werden, wodurch die HOCl-Bildung indirekt reduziert wird
- Schließlich ist evtl. auch noch die Sekretion von Halovibrin A und B zu nennen, die ADP-Ribosyltransferase Aktivität zeigen. Evtl. führt dies zu einem zumindest teilweisen Abschalten des oxidativen Bursts (für Ctx, einem ähnlichen Enzym aus V. cholerae wurde dies schon nachgewiesen)
- der zur Verfügung stehende Sauerstoff im Leuchtorgan absolut limitierend ist; er wird ja für die Lichterzeugung durch die, aber auch für die Atmung der Bakterien benötigt.
Es wurde vor einigen Jahren auch gezeigt, dass in den Tintenfischen Sepiola affinis und Sepiola robusta (beide im Mittelmeer), ein Gemisch aus Vibrio fischeri und Vibrio logei vorkommt, wobei die letztere Art immer überwiegt. Damit wurde zum ersten Male die Teilnahme der letzteren Bakterienart an einer Symbiose nachgewiesen und gleichzeitig gezeigt, dass die Leuchtorgane auch mehr als eine Art enthalten können. Ein Grund dafür könnte darin zu sehen sein, dass V. logei ab ca. 20° C nicht mehr gut leuchtet, während V. fischeri auch über 24° C leuchtet. Die Wassertemperaturen im Mittelmeer liegen ja durchaus für längere Zeit unter 20° C.
Vergleiche der Systeme von V. fischeri, V. harveyi und Gram-pos. Bakterien
Im Falle von Gram-pos. Bakterien wirken prozessierte Peptide, die wieder auf 2-Komponenten-Systeme verschaltet sind: ein prozessiertes Peptid wird ausgeschieden, über einen Membranständigen Rezeptor erkannt, der über ein 2-Komponenten-System entsprechende Target-Gene reguliert.